在发布第一份人类基因组草图20年后,Telomere-to-Telomere(T2T)联盟组装了世界上第一个完整基因组(T2T-CHM13)。T2T基因组完整表征了单个人类基因组所有序列,旨在提供对人类基因组更全面和准确的描述。以往的人类基因组组装通常在某些区域存在缺失、不连续或不准确的部分,而T2T基因组的目标是填补这些空白,实现从端粒到端粒的连续组装。
GRCh38是现有的人类基因组模版,在多数科学研究中被广泛应用(如:关联分析,疾病风险位点分析,演化分析等)。然而,现有的人类基因组模版(GRCh38)在组装中存在大量的未知序列且多数复杂区域组装并不完整准确。因此,T2T基因组相对于GRCh38基因组提供了更完整、更准确的基因组序列,具有更高的连续性和精确性。未来随着T2T基因组的进一步发展和广泛应用,我们需要更精准地理解这两个基因组组装版本之间的差异和优势。
2023年7月4日,上海交通大学毛亚飞课题组在Genome Biology发表题为Characterization of large-scale genomic differences in the first complete human genome的研究论文,比较分析了T2T-CHM13完整基因组与当前人类参考基因组模版(GRCh38)之间的大规模基因组差异,系统地表征了两个人类基因组组装之间的大型结构变异(≥10 kbp),通过新开发的结构变异分析工具网站(SynPlotter)验证238个基因组差异区域并发现了67个新鉴定的结构差异区域。
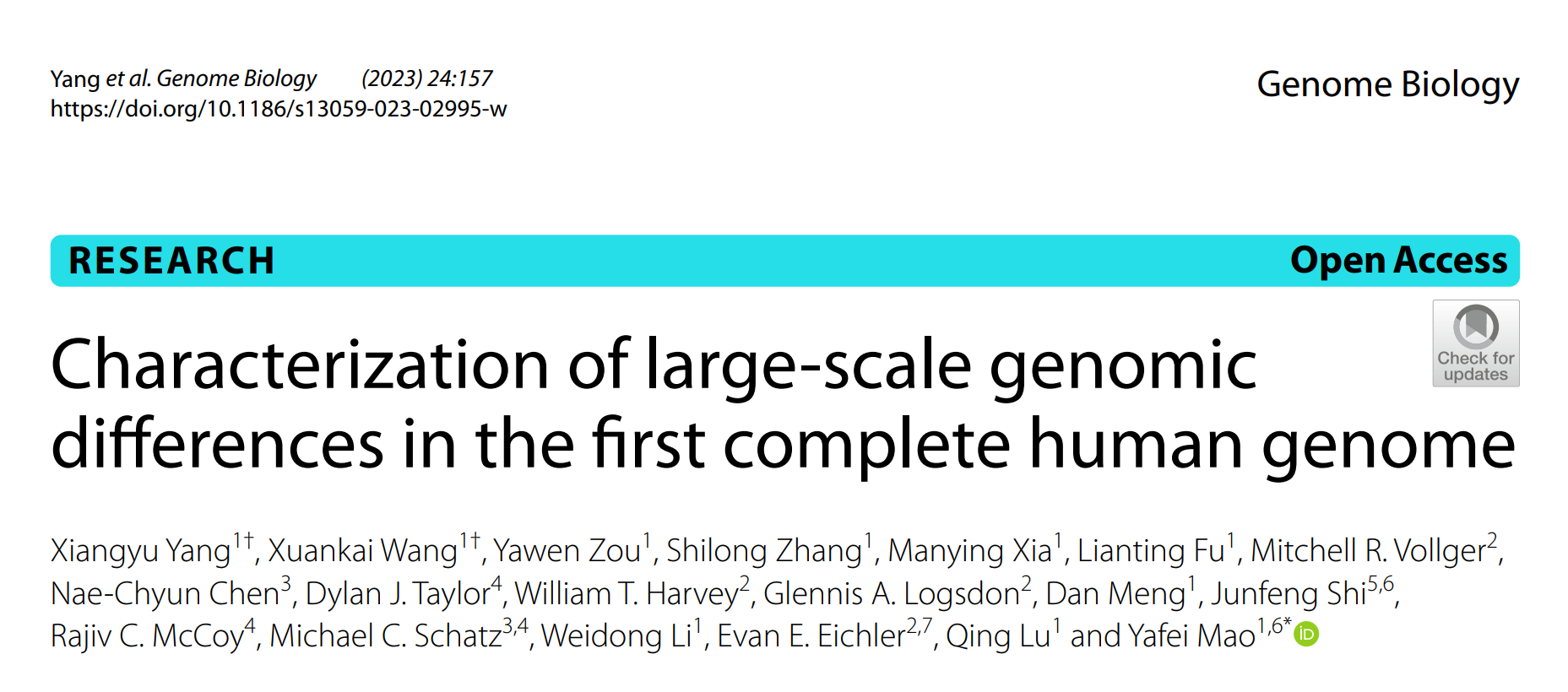
在本项研究中,毛亚飞团队使用了三种不同的结构变异分析方法来识别GRCh38和T2T-CHM13之间的结构变异(≥10 kbp),以确定基因组大规模差异区域并精确地锚定断点和结构类型。
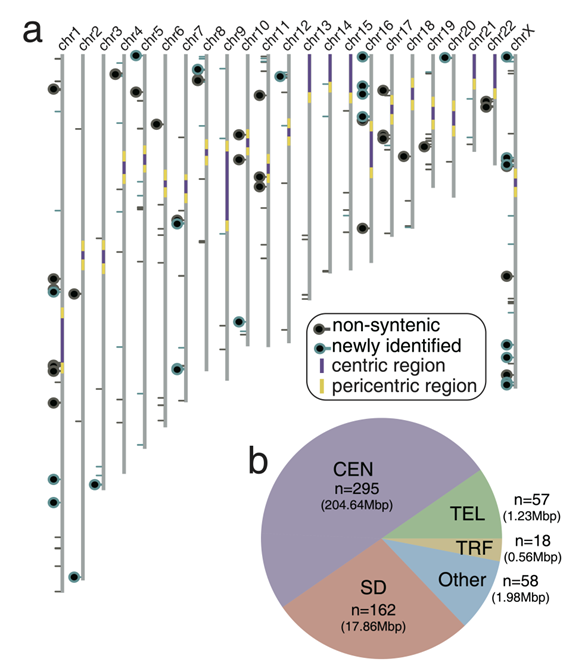
图1 GRCh38与T2T-CHM13基因组区域差异分析
该研究发现基因组差异区域中存在着许多与生理功能相关的基因,这些基因的差异与人类脑、免疫等相关疾病息息相关。研究团队着重分析了新鉴定出的基因组差异区域中KLRC基因簇,比较了人类群体间和非人灵长类(NHP)中该基因簇的差异。以KLRC2为例,从进化、种群分型、蛋白功能及结构等多个维度,探索KLRC2的重复和缺失机制及其生理功能。
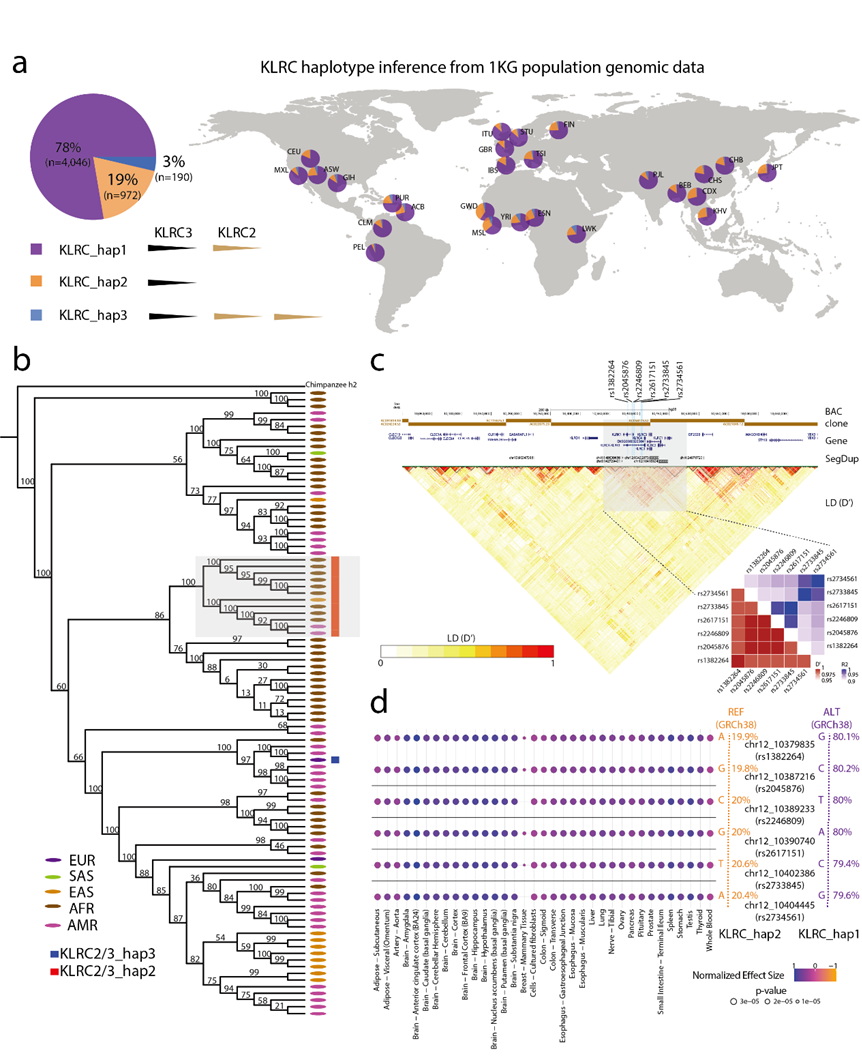
图2 人类KLRC2的结构和功能多样性
总的来说,该项研究对GRCh38和T2T-CHM13之间大规模差异基因组区域的结构和功能进行更全面和详细评估。该结果不仅有助于我们对基因组中复杂结构遗传多样性的认识,还提出了消除参考偏差来推动未来的科学研究。研究者认为该研究的新范式将在今后与HPRC (Human Pangenome Reference Consortium)、CPC (Chinese Pangenome Consortium)和Primate T2T(Telomere-to-Telomere Consortium)产生的遗传多样性的完整基因组结合,将有助于我们充分了解人类复杂基因组片段的多样性和功能,将极大扩展人们对复杂基因组片段的生物学认知。
本研究由上海交通大学Bio-X研究院毛亚飞实验室主导完成。上海交通大学Bio-X研究院助理研究员杨翔宇博士、硕士研究生王宣凯为本文共同第一作者。毛亚飞长聘教轨副教授为本文的通讯作者。该工作还得到了上海交通大学陆青研究员、李卫东教授,华盛顿大学Evan E. Eichler教授、约翰霍普金斯大学Michael Schatz教授等专家学者的大力支持;并得到上海市浦江人才计划,上海交通大学“交大2030”计划,国家自然科学基金青年项目等项目的资助。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-02995-w
参考文献:
1. Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. Initial sequencing and analysis of the human genome. Nature. 2001;409:860–921.
2. Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. The sequence of the human genome. Science. 2001;291:1304–51.
3. Nurk S, Koren S, Rhie A, Rautiainen M, Bzikadze AV, Mikheenko A, et al. The complete sequence of a human genome. Science. 2022;376:44–53.
4. Yang X, Wang X, Zou Y, Zhang S, Xia M, Fu L, et al. Characterization of large-scale genomic differences in the first complete human genome. Genome Biol. 2023:24, 157.
5. Wang T, Antonacci-Fulton L, Howe K, Lawson HA, Lucas JK, Human Pangenome Reference Consortium, et al. The Human Pangenome Project: a global resource to map genomic diversity. Nature. 2022 Apr;604(7906):437-446.
6. Gao Y, Yang X, Chen H, Tan X, Yang Z, Chinese Pangenome Consortium (CPC),et al. A pangenome reference of 36 Chinese populations. Nature. 2023 Jun 14.
7. Aganezov S, Yan SM, Soto DC, Kirsche M, Zarate S, Avdeyev P, et al. A complete reference genome improves analysis of human genetic variation. Science. 2022 Apr;376(6588):eabl3533.